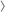FAQ
- Oligo
- Cloning
- Genecopoeia
- Genepharma
- 결제/기타
-
기본적으로 모든 주문건에 대해서 Polyacrylamide gel electrophoresis로 품질관리를 실시하고 있습니다.
50 mer 이하의 제품에 대해서는 MALDI-TOF/MS에 의한 질량 분석을 실시하고 있습니다. -
Oligonucleotide는 일반적으로 건조된 상태로 공급됩니다. 이것은 운송과정에서 노출될 수 있는 여러가지 환경적 요인으로 인한 oligo의 변성을 방지하기 위함입니다. 건조된 oligo는 때때로 용기 개봉 과정에서 손실될 수 있으니 잠시 원심분리한 후 개봉하시기 바랍니다. 특히 latex 장갑 등 정전기를 일으키는 착용물을 끼고 뚜껑을 개봉하면 건조된 oligo powder가 손실될 위험성이 높으니 특히 주의를 요합니다. Oligo 제품을 수령하신 후 용기에 기재된 양의 TE buffer (10 mM Tri-HCl pH8.0, 1 mM EDTA) 또는 멸균증류수에 녹여 100 uM stock 상태로 -20℃이하에 보관하시고, 사용시 적정 농도로 희석하여 사용하시기를 권장합니다. 수용액 상태에서 oligo는 멸균증류수보다 TE buffer가 안정하기는 하나 경우에 따라서 TE buffer 조성이 반응에 영향을 미칠 수 있으니 주의바랍니다. 수용액 상태의 Oligo를 반복적으로 동결/해동할 경우 변성될 위험성이 있으니 소량 분주하여 사용하시기를 권장합니다.
-
당사에서는 99 mer까지를 합성 한계로 정하고 있습니다. 그 이상의 길이를 의뢰하실 경우는 당사에 문의해 주시기 바랍니다.
-
Oligo 합성에 사용되는 Column내 CPG (controlled pore glass)의 양에 따라 합성되는 oligo의 합성수율은 달라집니다. 예를 들어 0.05 umole 스케일로 주문 하실 경우, 3'base가 최소 0.05 umole 이상 coupling 될 수 있는 양의 CPG가 충진된 column을 사용합니다. 이후 합성과정에서 매 cycle 다음 염기가 coupling 되지 못한 0~2%를 배제시키는 capping 과정에서 손실이 발생하고, 그 다음 정제공정상에서도 손실이 발생합니다. 따라서 0.05 unmole 스케일이란 합성 시 시작량을 의미하기 때문에 최종 양과 차이가 있을 수 있습니다. 또한 0.2 umole 스케일 주문시 0.05 umole 스케일로 주문하셨을 때와 최종 양이 반드시 비례 증가되진 않습니다.
-
Oligo size, coupling efficiency, base composition, 정제방법 등으로 인해 합성 수율은 달라집니다.
-
동일한 oligo를 합성한다해도 합성과정과 정제과정에서의 여러 가지 요인들로 인해 oligo의 합성 수율은 차이가 나게 됩니다.
-
Oligo의 50%가 그 상보서열에 결합해 있을 때의 온도를 의미하며, 이는 절대값이 아니고 농도 및 수용액상의 salt 농도 등에 따라 달라집니다.
-
Tm 값의 계산식은 여러 가지 종류가 있어 채용하는 계산식에 따라 값이 다릅니다. 간혹 고객님께서 사용하시는 계산식 program에 따라 당사의 계산과 다른 결과가 나오기도 합니다. 당사에서 사용하는 Tm calculator는 일반적으로 염기 개수에 일정 값을 곱하는 단순 계산식이 아닌, 염기 조성뿐만아니라 배열 순서에 따라 Tm 값이 달라지는 점을 고려하여 다음 계산식을 채택하고 있습니다. Tm 값은 이론적으로 계산된 값이기 때문에 실제 실험결과와 일치하지는 않습니다. 따라서 PCR 등에 oligo 사용시 실험적으로 최적 온도 적정이 필요합니다.
* 17 mer 이하 :
4×(G+C+R/2+Y/2+M/2+S+K/2+D/3+H/3+2B/3+2V/3+N/2)+2×
(A+T+R/2+Y/2+M/2+W+K/2+2D/3+2H/3+B/3+V/3+N/2+I+U)
* 18 mer 이상 :
60.8+41×{(G+C+R/2+Y/2+M/2+S+K/2+D/3+H/3+2B/3+2V/3+N/2)/염기수}-(500/염기수)
* Salt 농도 조건
0.0567(M)=0.05(KCl 농도)+0.01×0.67(Tris 농도)
50 mM KCl농도
10 mM Tris-HCl 농도(pH8.4-9.0 at 25℃) -
OPC (Oligonucleotide Purification Cartridge) 정제는 간이 컬럼 정제라고도 하며, 역상크로마토그래피 (reverse-phase chromatography) resin, Oligo R3가 충진된 column을 사용합니다. 합성이 끝난 Oligo는 정상적인 합성이 완료된 것과 coupling을 이루지 못해 capping된 불완전 산물이 혼합되어 있습니다.
합성 마지막 단계에서 DMT기가 결합된 정상적으로 합성이 완료된 oligo는 DMT기의 소수성으로 OPC resin에 결합하게 되고 나머지 불완전한 산물 및 부산물들은 column을 통과해서 빠져나갑니다. 그 다음 oligo에서 DMT기를 떼어내는 과정을 통하여 resin으로부터 oligo를 회수하게 됩니다. -
50 mer 이상의 긴 oligo 합성시 고순도 oligo를 필요로 할 때 PAGE정제를 시행합니다. OPC 정제 산물을 PAGE (Polyacrylamide Gel Electrophoresis)로 전개하여 주밴드만 분리하는 정제법으로 90% 이상의 고순도 oligo를 확보할 수 있습니다. 정제 과정에서 손실이 많아 최종 수득량이 다소 적을 수 있습니다.
-
50 mer 이하의 oligo 합성시 고순도 oligo를 필요로 할 때 RP-HPLC (Reverse-phase HPLC) 정제를 시행합니다. 기본 원리는 OPC와 유사하나, HPLC column을 사용하기 때문에 높은 분리능으로 90% 이상의 고순도 oligo를 확보할 수 있습니다. 특히 형광이 표지된 oligo 합성시 반드시 필요한 정제 공정입니다. 정제 과정상의 손실로 최종 수득량이 다소 적을 수 있습니다.
-
Oligo 합성 과정에서는 매 cycle당 약 0~2%는 coupling반응이 일어나지 않아 capping으로 제거됩니다. 따라서, oligo 길이가 길어지면 그만큼 합성 실패 percentage가 증가하게 됩니다. 99% 효율일 때 정상의 oligo와 잘못된 oligo의 혼합비율은 아래표와 같습니다.
Percentage Yield of Full-length and Failed Oligo by Length
참고로 표에서는 전체 합성효율에 영향을 미칠 수 있는 다른 화학적 요소를 배제한 것으로, 대표적으로 purine계 염기 (특히 'A')에서 base가 떨어져 나가는 "depurination" 현상입니다.Length(no. of bases) Product Failure 10 91 9 20 83 17 50 61 39 75 48 52 100 37 63
Depurination된 염기는 deprotection 단계에서 보통 제거되며 이를 "X elimination" 이라 합니다. Depurination의 비율은 매우 낮으나 올리고 길이에 따라 크게 늘어납니다. -
G가 4개이상 반복되면 guanine tetraplex 형태로 aggregation 될 수 있기 때문에 합성에 어려움이 있습니다. 당사에서는 오랜 합성 경험을 통해 얻은 기술로 연속 G가 포함된 oligo도 큰 어려움 없이 합성해 오고 있습니다. G가 반복되는 oligo 합성시 PAGE 및 HPLC가 어려운 문제가 있기 때문에 OPC 정제를 권장합니다.
-
인터넷 홈페이지의 주문란 (Onlinr Order → 서비스주문 → 로그인(회원가입필요) → Cloning)을 이용하시거나, 클로닝팀 메일(cloning@cosmogenetech.com)으로 cloning에 필요한 정보를 보내주세요
-
코스모진텍 홈페이지의 서비스가격 참고하시기 바랍니다.
( Service -> Cloning -> 결과확인 ) -
Plasmid DNA를 template으로 진행하는 cloning의 경우 약 3주 가량이 소요되며, cell, RNA, cDNA 및 gDNA를 template으로 진행하는 경우 4~5주가 소요됩니다. 진행 중간중간 결과보고를 드리니 그에 맞춰서 실험 준비를 하시면 됩니다.
-
Cloning service의 가격은 oligo(50mer 이하) 합성 가격 및 sequencing 비용이 모두 포함된 금액이며, 완료 시 유전자에 대한 full sequencing 결과와 cloning에 사용된 primer를 모두 제공해 드립니다.
-
Cloning하고자 하는 유전자에 대한 sequence 정보와 template DNA 및 목적하는 vector를 제공해 주셔야 합니다. 단 TA cloning의 경우에는 vector를 제공하지 않으셔도 됩니다.
-
일반적으로 연속된 6개의 염기까지는 quickchange method로 mutagenesis가 가능하고, 그 이상의 경우 overlap PCR method로 진행하는것이 효율적입니다. 자세한 내용은 상담을 통해 진행방법을 결정하실 수 있습니다.
-
Serum이 포함된 media를 사용할 경우 타사의 transfection reagent와 달리 jetPRIME®의 transfection 효율이 증가합니다. DNA/Reagent 복합체를 complete medium 내의 세포에 직접 첨가할 수 있습니다.
-
Polyplus의 transfection reagent 전제품은 antibiotics에 의해 transfection 효율에 영향을 받지 않습니다. 예를 들어 생산 과정에서 새로운 batch에 대한 jetPRIME®의 validation 프로토콜은 항상 serum과 antibiotics가 포함된 complete medium 조건에서 진행됩니다.
-
Cell density는 transfection 효율에 영향을 끼칩니다. jetPRIME®을 사용하여 최적의 transfection 효율을 얻기 위해 60%~80%의 confluency의 cell에 transfection할 것을 권장합니다. Culture plate 종류에 따라 권장하는 cell seeding 시의 cell수는 jetPRIME® transfection 프로토콜의 table 1을 참고해 주시기 바랍니다.
만약 cell을 오랫동안 culture 했다면 (20 passages 이상), 액체 질소에 보관중인 새로운 cell을 thawing하여 사용할 것을 권장합니다. -
Cell의 passage number는 transfection 효율에 많은 영향을 줍니다. 만약 cell을 오랫동안 culture 했다면 (20 passages 이상), transfection 효율이 감소할 것입니다. 그러므로 액체 질소에 보관중인 새로운 cell을 thawing하여 사용할 것을 권장합니다. 또한 cell을 thawing한 후 transfection 실험에 사용하기 전 최소 2회 이상 계대 배양할 것을 권장합니다.
-
여러 종류의 plasmid를 cotransfection 하고자 할 경우, well당 총 DNA양은 프로토콜에 표기된 DNA양을 초과해서는 안됩니다. 각각의 plasmid의 비율은 plasmid의 size, 종류 및 각각 plasmid의 예상 발현 수준에 따라 달라집니다. 각각의 plasmid의 양은 well당 총 DNA양의 최소 10% 이상은 되어야 합니다.
-
Transfection은 cell에 상당한 물리적 손상을 주게 됩니다. Cell viability를 향상시키기 위해 아래의 몇 가지 방법을 제안합니다.
- - Transfection 후 4시간 뒤에 media를 교체합니다.
- - DNA의 양을 줄입니다.
- - jetPRIME® reagent의 양을 줄입니다.
- - Complete medium에서 transfection을 수행합니다.
- - Transfection 후 24시간 뒤에 (48시간 대신) transfection efficiency을 분석합니다.
- - jetPRIME® buffer로 jetPRIME®과 DNA를 dilution 했는지를 확인합니다.
- - Plasmid에 endotoxin이 포함되어있는지의 여부를 확인합니다. (endotoxin free)
-
jetPRIME®는 classical media에서 virus를 생산하기 위한 최적의 reagent입니다. 통상 virus 생산에 사용되는 cells 즉, HEK-293 및 derivatives, CHO, VERO, WOP과 BHK cell에서 jetPRIME®을 사용했을 경우 transfection efficiency가 거의 90%에 달하므로 높은 virus titer를 얻을 수 있습니다.
(Dewannieux, M., Vernochet, C., Ribet, D., Bartosch, B., Cosset, F. L., Heidmann, T. (2011). The mouse IAPE endogenous retrovirus can infect cells through any of the five GPI-anchored Ephrin A proteins., PLoS Pathog 7, e1002). -
일반적으로 oligonucleotides (ODN)의 크기에 따라 INTERFERin® 또는 jetPEI®를 추천합니다. jetPRIME®는 oligonucleotides (ODN) transfection에는 적합하지 않습니다. oligonucleotides (ODN) transfection을 할 경우에는 technical support에게 문의 해주세요.
-
INTERFERin®은 siRNA transfection를 위한 제품이며, 적은 양 (1 nM)의 siRNA transfection시에도 효과적입니다. jetPRIME® 또한 siRNA transfection에 최적화되어 있으며, siRNA 최종 농도를 10~50 nM로 사용하면 됩니다.
-
T 및 B lymphocytes 등의 suspension cell은 어떤 종류의 chemical 기반의 transfection reagent를 사용하더라도 DNA transfection이 잘되지 않습니다. 그래서 suspension cells에 nucleic acid delivery를 하고자 할 경우에는 일반적으로 electroporation 또는 virus transduction 방법을 사용할 것을 권장합니다.
-
식물 세포는 셀룰로즈의 함량이 높은 막구조를 가지고 있어서 jetPRIME®/DNA 복합체가 cell안으로 들어가는 것이 차단됩니다. 심지어 셀룰로즈 세포벽이 제거된 protoplast 단계에서도 jetPRIME®/DNA 복합체는 cytoplasm으로 투과되지 못합니다.
-
HUVEC cells의 transfection을 위해 특이적으로 디자인한 jetPEI®-HUVEC을 사용할 것을 권장합니다.
-
Neurons은 배양시 세포분열을 하지 않기 때문에 transfection이 매우 어렵습니다. 그러나 jetPEI®는 in vitro 조건에서 primary neurons의 transfection에 성공적으로 사용된 사례가 있습니다. Polyplus는 primary neurons 세포의 DNA transfection을 위한 별도의 protocol을 가지고 있습니다. 더 자세한 내용은 technical support에게 문의해 주시기 바랍니다.
-
jetPRIME®을 사용하여 RAW 264.7 cell에서 성공적으로 DNA transfection 을 수행할 수 있으며, standard condition에서 대략 50%의 transfection 효율을 보입니다. Primary macrophages 또는 monocyte/dendritic cell-derived macrophages로 DNA transfection을 할 경우에는 jetPEI®-macrophage 제품을 선택하시면 됩니다. 자세한 정보는 아래의 링크에서 확인하실 수 있습니다.
(http://www.polyplus-transfection.com/products/jetpei-macrophage/) -
jetPRIME®는 Polyplus-transfection에서 새로 개발한 고유의 cationic polymer-based molecule입니다.
-
jetPRIME®은 DNA와 함께 positive charge의 복합체를 형성합니다. 이 복합체는 endocytosis 기전을 통해 세포 내로 들어오게 됩니다. DNA는 proton sponge mechanism을 통해 endosome에서 cytosol로 이동합니다. Cytoplasm 내의 plasmid는 nuclear envelope이 사라지는 mitosis 단계에서 대부분 nucleus에 도달하게 됩니다.
-
jetPRIME®은 매우 안정적인 분자입니다. jetPRIME®과 buffer는 상온으로 납품되지만, 장기간 안정한 상태를 유지하기 위해서는 4℃에서 보관해야 합니다.
-
jetPRIME®은 매우 안정한 분자입니다. 안정성 테스트 결과, jetPRIME®은 50°C에서 48시간 보관 후에도 4℃에서 보관하는 경우와 유사한 transfection efficiency을 나타냈습니다.
-
jetPRIME®은 의도치 않은 동결/해동 과정에도 transfection efficiency가 거의 변하지 않습니다. 그러나 장기 보관을 위해서는 제품의 장기간 안정성을 보장하는 4°C에서 보관해야 합니다.
-
적절한 상태로 보관되었다면, jetPRIME® cat#114-01 (0.1ml) 제품은 6개월간 안정합니다. 다른 사이즈의 jetPRIME® 제품은 최소 1년 이상 안정적으로 사용할 수 있습니다.
-
Ratio는 DNA 양 (㎍)에 대해 사용하는 jetPRIME® 부피 (㎕)의 비율입니다. 예를 들어 1:2 DNA to jetPRIME® ratio는 1 μg의 DNA 당 2 μl의 jetPRIME®의 비율로 사용하는 것을 의미합니다.
-
Transfection 효율을 높이는 최적화의 첫번째 과정은 DNA 양을 1.5배까지 증가시키는 것입니다. 또한 DNA to jetPRIME® ratio를 높이는 것을 권장합니다 (1 μg DNA당 2~3 μl jetPRIME® 비율). 또한 culture plate를 210 X g에서 5분간 centrifugation을 진행할 수도 있습니다.
-
jetPRIME®은 A549, MCF7, U-2OS, NIH-3T3, B16-F10, Caco-2...의 다양한 cell line에 최적화된 프로토콜을 제공합니다. 그러나 특정한 cell lines의 경우 더 높은 transfection 효율을 얻기 위한 별도의 프로토콜을 보유하고 있습니다. 이러한 실험 조건들은 Polyplus-transfection Database에서 확인할 수 있습니다. (http://www.polyplus-transfection.com/resources/cell-transfection-database/)
-
jetPRIME®은 HEK-293과 HeLa cell의 DNA transfection에 매우 효율이 높습니다. 그러므로 DNA양을 줄이고 (6-well 기준 well당 1 μg을 0.5 μg으로), DNA to jetPRIME® ratio를 1:2로 진행할 것을 권장합니다.
-
일반적으로, DNA의 양은 세포의 총 수에 비례해서 증가해야 합니다. 사용되는 culture dish의 크기에 따라 필요한 jetPRIME®과 DNA의 양이 jetPRIME® 프로토콜 (table 2)에 기재되어 있습니다.
-
jetPRIME®을 사용할 경우에는 형성된 지 15~30분 정도의 jetPRIME®/DNA 복합체에서 최적의 transfection efficiency를 얻을 수 있습니다. 그러므로 incubation 시간이 많이 필요한 High Throughput Screening (HTS) 실험에 적용할 경우에는 복합체 형성 후 4시간까지 안정한 상태를 유지할 수 있는 jetPEI®를 사용할 것을 권장합니다.
-
이 경우 plasmid의 크기에 제한이 없습니다. 하지만, 동일한 양의 DNA의 경우에 size가 큰 plasmid가 작은 것보다 gene copy number가 더 적다는 것을 염두에 두고 실험 디자인을 해야 합니다.
-
홈페이지상의 Polyplus-transfection Database에서 검색을 통해 다수의 cell line에 대한 별도의 프로토콜을 찾을 수 있습니다.
(http://www.polyplus-transfection.com/resources/cell-transfection-database/) -
INTERFERin®은 cell culture시 mammalian cell에 siRNA를 전달하기 위해 개발된 새로운 방식의 nonliposomal cationic amphiphile transfection reagent입니다. INTERFERin®을 사용하여 1 nM의 siRNA에서 90%이상의 silencing efficiency을 얻을 수 있습니다.
-
INTERFERin®은 HeLa, HEK-293, A549, MDA-MB-231, BHK-21, CHO 및 OVCAR-3 등의 다양한 adherent cell 및 non-adherent cell line에서 성공적으로 테스트 되었습니다. 또한 INTERFERin®은 primary human fibroblast 및 primary human hepatocytes 등의 다양한 primary cells에서도 사용할 수 있습니다. 자세한 내용은 Polyplus-transfection Database에서 확인할 수 있습니다.
(http://www.polyplus-transfection.com/resources/cell-transfection-database/) -
다양한 종류의 분자들이 miRNA로 부정확하게 불려지고 있습니다. miRNA를 아래와 같이 구분할 수 있습니다.
- - mimic miRNA : 이것은 siRNA와 동일하지는 않지만 구조적으로 매우 유사하며, INTERFERin®으로 transfection할 수 있습니다.
- - plasmid encoding for miRNA : 이것은 DNA이므로 jetPRIME®으로 transfection할 수 있습니다.
- - antimiR : 이것은 세포 내에 존재하는 microRNA에 특이적으로 결합하여 그 기능을 저해하도록 디자인된 single-stranded nucleic acid이며, INTERFERin®으로 transfection할 수 있습니다.
-
INTERFERin®은 in vitro transfection 전용 reagent입니다. 동물에서 siRNA delivery 실험을 하고자 한다면, 다양한 종류의 동물에 siRNA 및 oligonucleotides의 delivery가 가능한 in vivo-jetPEI®를 사용할 것을 권장합니다. 자세한 정보는 홈페이지를 참고해 주시기 바랍니다.
(http://www.polyplus-transfection.com/products/in-vivo-jetpei/) -
INTERFERin®은 반드시 4°C에서 밀봉한 채로 보관해야 합니다.
-
INTERFERin®은 보관 온도에 따라 효율에 차이가 발생하므로 절대 동결되면 안됩니다. 만약 동결되었다면, 버리고 새 제품을 사용해야 합니다.
-
적절한 보관 조건을 유지하는 경우, INTERFERin® cat#409-01 (0.1 ml)은 6개월간 안정한 상태를 유지합니다. 다른 사이즈의 INTERFERin®은 최소 1년이상 안정한 상태를 유지합니다.
-
PULSin™은 항체와 펩타이드 등의 많은 protein들과 positive charged coat를 갖는 복합체를 형성할 수 있는 cationic amphiphile molecule을 함유하고 있습니다. 이러한 PULSin™/proteins 복합체는 실제 모든 세포막에 존재하는 syndecans에 결합하여 세포 안으로 들어가며, 이후 endosome에서 cytoplasm으로 단백질이 이동하게 됩니다.
-
HeLa 및 NIH-3T3 등의 established cell line 및 Jurkat 등의 suspension cells, 그리고 human keratinocytes와 같은 primary cell 등 다양한 type의 cell에서 PULSin™ kit의 positive control 단백질인 r-phycoerythrin의 PULSin™-mediated delivery가 성공적으로 테스트되었습니다. 자세한 내용은 Polyplus-transfection Database에서 확인할 수 있습니다.
(http://www.polyplus-transfection.com/resources/cell-transfection-database/) -
PULSin™을 사용하여 40~250 kDa 크기의 생리학적 pH에서 negative charge (단백질 pI 범위 : 0 ~ 7.4)를 갖는 단백질을 성공적으로 delivery할 수 있습니다. 또한, anti-giantin, anti-vimentin, anti-tubulin 등 세포 구성 성분을 인식하는 다양한 antibodies 를 delivery한 후에도 계속 target 단백질을 인식할 수 있음을 확인하였습니다. Peptides 역시 성공적으로 delivery할 수 있으며, 보다 자세한 사항은 홈페이지를 참고해 주시기 바랍니다.
(http://www.polyplus-transfection.com/transfection-reagents/protein-delivery-reagent-pulsin/) -
PULSin™으로 운반되는 antibodies, proteins 또는 peptides는 세포질로 이동하여 PULSin™/protein 복합체에서 방출됩니다. 50 kDa 이하의 단백질의 경우는 nucleus로 diffusion될 수도 있습니다. 큰 size의 단백질을 nuclear targeting하려면 단백질 내에 nuclear localization signal (NLS)을 가지고 있어야 합니다.
-
PULSin™은 보관 온도에 따라 효율의 차이가 발생하므로 절대 동결되면 안됩니다. 만약 동결되었다면, 버리고 새 제품을 사용해야 합니다.
-
jetPEI® (in vitro 전용)와 in vivo-jetPEI®는 기본적으로 동일한 활성 분자를 기반으로 하고 있지만, 각각의 적용 목적에 따라 특이적으로 개발되었습니다. 그러므로 이들의 조성, 농도, 순도 및 검증테스트 방법에는 서로 차이가 있습니다. (in vivo-jetPEI®의 경우 endotoxin-free 조건에서 mouse에서의 in vivo gene delivery에 대해 검증테스트를 진행합니다.)
-
in vivo-jetPEI®은 매우 안정한 화합물로 상온 상태로 배송됩니다. 하지만 장기보관을 위해서는 제품 수령 후 -20°C에 보관하는 것을 권장합니다.
-
in vivo-jetPEI®를 사용하면 DNA, siRNA, miRNA mimic, antimiR, oligonucleotides 등 다양한 종류의 nucleic acids에 대해 in vivo delivery가 가능합니다. 자세한 내용은 Polyplus-transfection Database에서 확인할 수 있습니다.
(http://www.polyplus-transfection.com/resources/cell-transfection-database/) -
in vivo-jetPEI®는 mouse, rats, mosquitoes, shrimps, guinea pig, macaques 등 다양한 종의 동물에서 테스트 되었습니다. 더 자세한 내용은 technical support에게 문의해주시기 바랍니다.
-
in vivo-jetPEI®은 전신 또는 국소 부위로의 nucleic acid delivery에 모두 적합합니다. tail vein (꼬리 정맥) and retro-orbital (안와정맥) 등으로의 intravenous injection (정맥 주사), intraperitoneal injection (복강 주사), intratumoral injection (종양 내 주사), intracerebral injection (뇌내 주사), intranasal instillation (비강내 점적) 등 다양한 투여 경로에 대한 테스트가 성공적으로 진행되었습니다. 자세한 내용은 technical support 에게 문의하시거나 홈페이지 내의 Application Note (Publications by Administration route)를 참고해주시기 바랍니다.
-
in vivo-jetPEI®은 폐에 nucleic acid delivery를 하기에 적합한 제품입니다. 실제로 폐는 in vivojetPEI ®/nucleic acid 복합체가 systemic delivery시 가장 많이 targeting되는 장기입니다.
-
Delivery 과정은 표적 장기에 따라 매우 상이할 수 있습니다. 어떤 투여 경로를 선택하느냐에 따라 in vivo-jetPEI ® 매개의 유전자 발현이 뇌, 간, 췌장, 비장, 신장, 심장, 방광, 피부, 망막 등의 장기에서 각각 다르게 관찰될 수 있으므로, 자세한 정보가 필요한 경우 technical support에게 문의해주시기 바랍니다.
-
jetPEI® transfection reagent는 자동화 또는 수동의 HTS를 수행하는데 가장 최적화되어 있으며, Reverse, Batch, Forward 세가지 프로토콜을 모두 사용할 수 있습니다. jetPEI® reagent로 일관된 반복 실험 결과를 얻을 수 있으며, DNA/jetPEI® 복합체는 4시간까지 안정한 상태를 유지합니다. 보다 자세한 내용은 홈페이지를 참고하시기 바랍니다.
(http://www.polyplus-transfection.com/products/jetpei/) -
INTERFERin®-HTS는 HTS 환경에서 최대의 gene silencing 효율을 얻을 수 있도록 특별히 개발된 새로운 개념의 siRNA 전용 transfection reagent로 최고의 재현성과 소량의 reagent를 사용하므로 향상된 cell viability 를 유지할 수 있습니다. INTERFERin®-HTS는 serum 및 antibiotics가 포함된 media에서 사용이 가능하며 합리적인 가격과 취급 용이성을 가지고 있습니다. 96-well과 384-well plates에서 forward 또는 reverse transfection을 진행할 수 있는 실험 프로토콜을 제공합니다. 자세한 내용은 홈페이지를 참고하시기 바랍니다.
(http://www.polyplus-transfection.com/products/interferin-hts/) -
jetPEI®는 linear polyethylenimine (PEI)으로 구성되어 있으며, 분자량은 polyplus의 고유 정보로 공개할 수 없습니다. Polyplus는 in vitro 조건에서 가장 세포 독성이 낮으면서 최적의 DNA delivery 효율을 갖는 linear PEI를 선택하기 위해 수많은 최적화 실험을 진행하여 jetPEI®를 제품화했습니다.
-
jetPEI®은 HTS 이외에 in vitro DNA transfection 등의 용도로도 사용할 수 있습니다. 또한 primary neuron에 jetPEI® 매개의 DNA transfection이 가능한 별도의 프로토콜을 개발했습니다. 자세한 정보가 필요한 경우 technical support에게 문의해주시기 바랍니다.
-
FectoPRO®은 suspension cell에서의 바이러스 생산에 완벽하게 최적화 되어있습니다. PEIpro®는 adherent cell에서의 바이러스 생산에 더 적합합니다. 두 제품 모두 치료용 바이러스 생산에 적합하도록 광범위한 품질 검증을 거쳐 생산됩니다.
-
FectoPRO® 및 PEIpro®는 모두 biomanufacturing을 위해 사용할 수 있습니다. FectoPRO®는 suspension 상태로 배양하는 CHO 또는 HEK-293 cell에서 매우 높은 단백질 수율을 달성하기에 특히 적합합니다. 그러나 대용량 또는 초대용량의 scale로 실험을 진행할 경우에는, HEK-293 세포에서 매우 효율이 높고 상대적으로 경제적인 PEIpro®를 선택할 수도 있습니다.
-
FectoPRO®는 FectoPRO® booster 없이도 사용할 수 있지만, 더 적은 양의 DNA를 사용하여 높은 단백질 수율을 달성하고자 한다면, booster를 사용할 것을 권장합니다.
-
FectoPRO®는 예기치 않은 동결/해동 과정에도 transfection efficiency를 유지할 수 있는 안정적인 제품이지만, 오랜 기간 동안 안정한 상태를 보장하기 위해서는 4℃에 보관하는 것을 권장합니다.
-
PEIpro®는 linear polyethylenimine (PEI)으로 구성되어 있으며, 분자량은 polyplus의 고유 정보로 공개할 수 없습니다. Polyplus는 in vitro 조건에서 가장 세포 독성이 낮으면서 최적의 DNA delivery 효율을 갖는 linear PEI를 선택하기 위해 수많은 최적화 실험을 진행하여 PEIpro®를 제품화했습니다.
-
PEIpro®은 1 ㎎/ml의 높은 순도와 품질의 linear PEI의 수용액으로 제공됩니다.
-
PEIpro®는 예기치 않은 동결/해동 과정에도 transfection efficiency를 유지할 수 있는 안정적인 제품이지만, 오랜 기간 동안 안정한 상태를 보장하기 위해서는 4℃에 보관하는 것을 권장합니다.
출처: http://www.polyplus-transfection.com/technical-resources/faq/
-
Clone이 제작되면, GeneCopoeia는 PCR을 이용한 size 분석, ORF full sequencing, Restriction Enzyme 처리를 통한 plasmid clone의 정확성 여부를 확인하는 엄격한 품질관리 절차를 거쳐 정확한 clone을 고객에게 제공합니다.
-
Empty vector 구매 가능하며, 연구 시 negative control로 사용할 수 있습니다. ORF clone과 함께 구매할 때 좀 더 저렴하게 구매하실 수 있으며 vector마다 금액은 상이할 수 있으니 학술지원팀에 문의하여 주시기 바랍니다.
-
시퀀스의 차이는 아래와 같은 두 가지의 잠재적인 요인에 의해 발생할 수 있습니다.
- 1) 자연적으로 발생할 수 있는 polymorphisms, alternative splicing, annotation 업데이트에 기인한 동일한 유전자의 version 및 copy의 불일치, Sequencing 에러 및 분석 오류
- 2) PCR cloning 과정에서 발생할 수 있는 시퀀스 오류
-
자연적으로 발생할 수 있는 protein-coding sequence와 NCBI reference sequence간의 차이로 인해, 당사는 OmicsLink™ ORF clone sequence와 NCBI reference sequence의 100% 일치를 보장하지 않습니다. 당사는 OmicsLink™ ORF expression clone sequence가 point mutation에 의해 non-stop codon이 stop codon으로 변환되어 발생할 수 있는 translation termination mutation 및 deletion, insertion등 인위적인 frame-shift에 의해 발생하는 변이가 없음을 보장합니다. 이러한 당사의 정책이 PCR에 의해서는 거의 일어나지 않는 자연적으로 발생할 수 있는 polymorphisms 및 mutation을 보장하는 것은 아닙니다. 만일 다른 cDNA library로부터 특정한 version의 gene ORF clone을 원하는 경우, 당사는 특정 version의 clone 제작을 위해 최선의 노력을 다하겠지만, 성공을 보장할 수는 없습니다.
-
pReceiver-B01 및 pReceiver-WG02/WG03/WG04/WG05/WG09/WG16의 모든 expression clones은 E. coli에서 expression test 또는 Roche사의 cell free protein production system을 이용하여 테스트 하였습니다. 이외의 다른 특성의 clone 및 다른 host cell system에 대한 expression clone들은 단백질 발현 테스트가 진행되지 않았습니다.
-
Knockdown 효율은 transfection 효율이 80% 이상일 경우 측정해야 합니다. Transfection 효율은 Vector 내의 reporter gene의 발현으로 확인할 수 있습니다. Transfection한 cell로부터 확보한 RNA를 사용하여 quantitative RT-PCR(qPCR)로 유전자 발현량의 감소 수준을 측정할 수 있습니다. 그러나 shRNA construct에 의한 silencing 효율을 측정하는 데에는 RT-qPCR 보다는 Western Blot을 추천하며, 유전자 발현량은 반드시 scrambled control clone 과 shRNA clone을 각각 transfection한 cell을 비교하여 확인해야 합니다.
-
GeneCopoeia는 발현 cassettes안의 shRNA 서열이 target 유전자와 동일함을 보장합니다. 만약 4개 construct 중 그 어떤 것도 qRT-PCR 상에서 70% 이상의 knockdown 효율을 보여주지 않으며 이러한 knockdown 효율이 construct가 원인이라고 판단되는 경우, specific 유전자를 targeting하는 새로운 4개 clone을 무료로 제공해드립니다. 이를 위해 고객은 control clone과 함께 확인한 quantitative RT-PCR(qPCR), 혹은 Western blot 실험에 대한 결과를 제공하여야 합니다.
-
TALE과 CRISPR은 genome editing을 시작하기 위해서 특이적으로 디자인된 target sequence를 인식합니다. 두 시스템의 가장 큰 차이점은 target sequence를 인식하는 방식입니다.
CRISPR은 20 nucleotide target sequence와 동일한 single guide RNA (sgRNA)를 사용합니다. Target sequence는 Protospacer Adjacent Motif (PAM)으로 알려진 3 nucleotide (5’-NGG-3’) 바로 뒤에 존재해야 하며, sgRNA는 원하는 site로 Cas9 nuclease를 guide하게 됩니다. CRISPR system을 사용할 경우에는 sgRNA sequence를 디자인해야 합니다.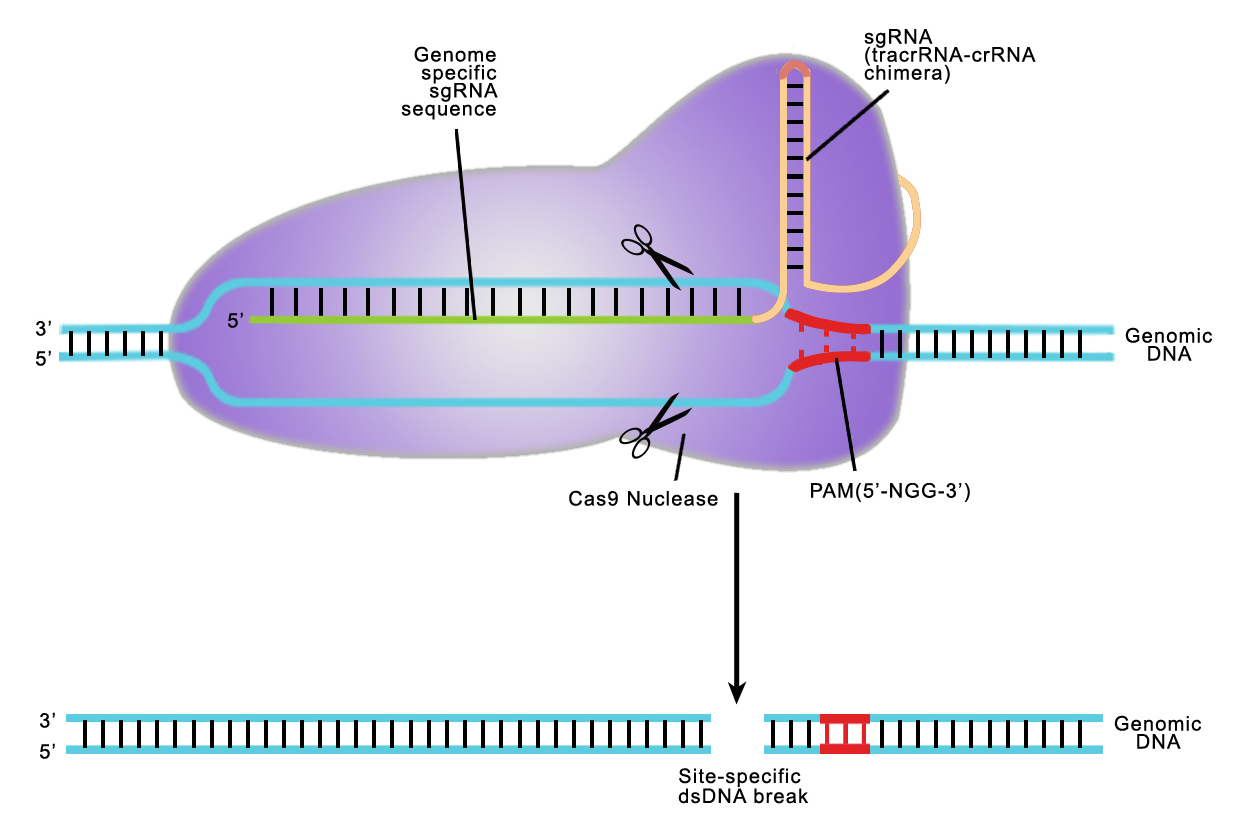 Figure Mechanism of CRISPR-Cas9:sgRNA target recognition and cleavage
Figure Mechanism of CRISPR-Cas9:sgRNA target recognition and cleavage
TALE은 연속적으로 이어진 34개의 amino acids로 구성된 repeats 내의 variable amino acid를 사용하여 target sequence를 인식할 수 있는 단백질입니다. 이러한 variant amino acids를 Repeat Variable Diresidues (RVDs)라고 합니다. 각각의 repeat는 1 RVD를 포함하며 각각의 RVDs는 1개의 특이적 nucleotide (A, T, G, C)를 인식할 수 있습니다.
 Figure Top : Schematic representation of a TAL effector. Bottom : Typically-used RVD recognition code
Figure Top : Schematic representation of a TAL effector. Bottom : Typically-used RVD recognition code
TALE은 nuclease 또는 transcriptional modulator 등의 genome editing motifs와 fusion되어 있습니다. TALE 기반의 system을 사용할 경우에는 target sequence를 정확히 인식할 수 있는 순서로 RVDs를 조립해야 합니다.
GeneCopoeia의 TALEN과 CRISPR sgRNA는 기본적으로 coding region의 앞쪽 부분을 targeting하여 해당 유전자로부터 파생될 수 있는 이미 알려진 또는 예측되는 transcript variants들을 모두 Knock-out 할 수 있도록 디자인 됩니다. -
TALEN과 CRISPR 시스템은 각기 장단점이 존재하며, 어느 system을 사용하는지의 선택은 실험자가 달성하고자 하는 연구목적에 달려 있습니다.
CRISPR은 TALEN보다 Gene을 editing하는 효율이 더 높습니다. 또한 DNA methylation 여부와 상관없이 작동하며, multiplex 실험이 훨씬 더 용이합니다. 이러한 부분을 TALEN에 비해 CRISPR이 가지고 있는 장점이라고 할 수 있습니다.
반면, Cas9 nickase를 포함하는 CRISPR의 경우는 TALEN에 비해 off-target site를 modification하는 성향이 더 강하므로, 연구자의 실험 목적 및 실험 적용에의 영향 여부에 따라 문제가 될 수도 있습니다. -
만약 단순히 human 또는 mouse 유전자 knockout을 하고자 할 경우, 연구자는 GeneCopoeia website에서 TALEN/CRISPR 검색 page에서 원하는 유전자를 찾고, 연구자의 system에서 작동할 수 있는 적합한 clone을 선별하여, GeneCopoeia 국내 대리점인 코스모진텍을 통해 견적 및 주문을 할 수 있습니다. 또한 검색 결과 page에서 knock-out에 사용할 수 있는 donor clones도 함께 주문할 수 있습니다. 만약 point mutation 등의 다른 application으로 실험해야 한다면, GeneCopoeia에 해당 내용을 요청하여 별도의 견적서를 받을 수 있습니다.
-
GeneCopoeia™은
- 1) 40,000 종 이상의 human 및 mouse ORF expression clones, human, mouse, rat 및 다른 동물의 genome-wide target genes에 대한 small hairpin RNAi (shRNA), 알려진 모든 human, mouse, rat miRNA에 대한 miRNA inhibitor clones
- 2) 20,000 종 이상의 human promoter reporter clones 및 18,000 종 이상의 mouse promoter reporter clones
- 3) Human 및 mouse의 CRISPR sgRNA clones 등
Lentiviral expression construct는 full-length sequencing, restriction enzyme digestion 및 gene-specific과 vector-specific primer을 사용한 PCR-size validation 을 통해 검증됩니다. GeneCopoeia™ EndoFectin Lenti Reagent (Cat No. EFL1001-01), TiterBoost reagent, 293Ta lentiviral packaging cell line (Cat No. CLv-PK-01) 와 mycoplasma detection kit을 함께 사용했을 경우, GeneCopoeia™ lentiviral product는 높은 viral titer를 얻을 수 있으며, bacteria, fungi 및 common mycoplasma의 오염이 없음을 확인할 수 있습니다. -
Lentivirus는 세포 독성을 가지고 있습니다. 필요 이상으로 많은 양의 lentivirus를 첨가할 경우 또는 infection 시간을 지나치게 오래 유지할 경우 연구자의 중요한 세포에 손상을 야기할 수 있습니다. 이러한 경우에는 multiplicity of infection (MOI)를 낮은 수준으로 조정해야 합니다. 우리는 transduction후 4~8시간 경과후에 culture media를 새로운 complete media로 교체할 것을 권장합니다 (transduction 후 12시간을 넘기지 않음).
-
10개의 사람 조직 total RNA 시료에서 얻어진 reverse transcript products인 cDNA pool이 검증을 위한 template로 사용됩니다. qPCR은 0.2 μM primer와 GeneCopoeia All-in-One™ qPCR Mix를 사용하여 수행됩니다. GeneCopoeia All-in-One™ qPCR pimers (또는 GeneCopoeia All-in-One™ miRNAqPCR primers)는 targeted gene과 miRNA에 대해 정확한 size의 단일 amplicon 생성 및 single dissociation curve peak 생성 여부 확인을 통해 검증됩니다.
-
실험 과정은 크게 두 가지 과정으로 나눌 수 있습니다.
첫번째 과정은 polyA polymerase를 사용하여 miRNA의 3’ 말단에 PolyA tail을 붙이는 것이며, 동시에 oligo(dT) primer와 MMLV reverse transcriptase를 사용하여 miRNA를 polyA tail 위치로부터 reverse transcription합니다.
두번째 과정에서 miRNA 특이적인 forward primer와 Universal Adapter primer를 PCR primer pair로 SYBR® Green이 포함된 qPCR master mix과 함께 Real Time PCR 을 수행하여 reverse transcribed miRNA를 특이적으로 검출하게 됩니다.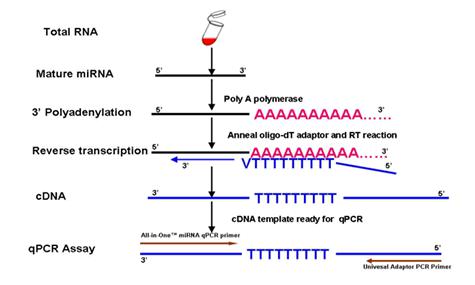 Figure 1. Experimental design used by the GeneCopoeia All-in-One™ miRNAqRT-PCR Detection Kit
Figure 1. Experimental design used by the GeneCopoeia All-in-One™ miRNAqRT-PCR Detection Kit
-
GeneCopoeia All-in-One™ miRNA primers와 함께 사용했을 경우, GeneCopoeia All-in-One™ miRNAqRT-PCR Detection Kit은 최소 20 pg의 total RNA 또는 10 pg의 small RNA에서 miRNA를 검출할 수 있습니다.
-
이 Array system을 사용하면, primer 설계와 유전자 및 miRNA의 검증에 드는 시간이 획기적으로 감소합니다. 또한 유전자 또는 miRNA의 실험 batch 당 qPCR 검출이 동시에 96-well 또는 384-well plate에서 가능합니다.
이것으로 실험의 단순화와 실험 속도 증가가 가능하게 됩니다.
-
코스모진텍 홈페이지의 online order를 통해 주문 가능하며, (Onlinr Order → 서비스견적/주문 → 로그인(회원가입필요) → RNAi) 학술팀(02-465-6217)으로 문의하셔도 됩니다.
-
GenePharma 제품은 OD 단위로 판매되며 1 OD = 2.5 nmole 입니다.
-
OD는 농도가 아니라 Quantity 입니다. siRNA의 경우 20uM 의 stock solution을 제조하기 위해 1 OD 당 125ul의 DEPC-treated water 를 첨가하시면 됩니다. (20uM = 20 pmole/ul)
-
Sequence 길이에 상관없이 주문하시는 OD에 따라서만 가격이 정해집니다.
-
dTdT는 siRNA의 stability 및 target mRNA와의 binding efficiency를 높여줍니다. chemical modified siRNA (2'-OMe or 2'-Fluoro)도 stability 및 target mRNA와의 binding efficiency를 높여줍니다.
-
주문시 custom siRNA sequence의 3' end에 dTdT 라고 써주시거나, remark 란에 dTdT를 붙여달라고 요청하시면 됩니다. dTdT에 대한 별도의 추가비용은 없습니다.
-
GenePharma는 60여종의 Target gene에 대한 Validated siRNA를 제공하고 있습니다. 리스트는 brochure를 참고해 주십시오 .
-
Target gene 에 대한 Accession no. 또는 Gene ID 를 메일(support@cosmogenetech.com) 또는 "online order" 를 통해 알려주시면, siRNA set 종류에 따라, 3개 또는 4개의 candidate siRNA 를 design 해드립니다. design 비용은 무료입니다.
-
pre-designed siRNA set A 는 하나의 target gene 에 대해 3개의 siRNA candidates (각 2OD), set B는 4개의 siRNA candidates (각 4OD)가 제공되며, 모든 Pre-designed siRNA set에는 Negative Control (1 OD), FAM-labeled Negative Control (1 OD), Positive Control (GAPDH, 1 OD)이 포함되어 있습니다.
-
Custom siRNA 경우는 guarantee가 되지 않으며, Pre-designed siRNA set의 경우 50% 이상의 transfection efficiency 조건에서 최소 1개의 siRNA candidates에서 70% 이상의 Knock-down efficiency를 guarantee 해드리며, 그렇지 않을 경우, 1회에 한해 replacement 해드립니다. 이 경우 FAM-labeled Negative Control에 대한 세포 이미지 사진 및 qRT-PCR 또는 western blotting 결과를 첨부해주셔야 합니다.
-
mature ID를 제공해 주시면 됩니다. (예: has-miR-21)
-
아래 글자를 누르시면 library 리스트파일(엑셀파일)을 다운로드 받으실 수 있습니다. miRNA Library 다운받기 (계속 업데이트되며, 리스트에 없는 mature ID의 제품도 주문 가능합니다.)
-
네, 가능합니다. Negative or Positive control siRNA, Negative or Positive miRNA mimics / inhibitor를 별도 구매하실 수 있습니다.
-
Control 제품군들은 판매 단위가 1 OD 입니다. 많은 양을 원하실 경우 주문 수량을 늘려서 주문하시면 됩니다.
-
Target gene에 대한 Accession no. 를 알려주시면 됩니다. target gene 에 대해 4개의 designed shRNA Plasmid Vectors 및 선택하신 vector에 해당하는 Negative, Positive Control Vector를 제공해 드립니다.
-
GenePharma의 shRNA는 다음 8종의 vector로 제작 가능하며 가격은 동일합니다.
(pGPU6, pGPH1, pGPU6/Neo, pGPH1/Neo, pGPU6/Hygro, pGPH1/Hygro, pGPU6/GFP/Neo, pGPH1/GFP/Neo)
다른 종류의 vector를 원하실 경우, 코스모진텍의 Cloning 서비스를 통해 shRNA clone을 제작할 수 있습니다. -
GenePharma의 RNAi 서비스는 수시발주로 진행되며, 배송 기간은 Custom siRNA 및 miRNA mimics 및 inhibitor는 10 OD 이하의 경우 영업일 기준 7일, pre-designed siRNA set는 3주, shRNA plasmid vector는 4주 소요됩니다.
-
요청서류와 방문 가능한 날짜를 알려주시면 담당영업사원의 방문을 통하여 처리하여 드리겠습니다.
-
계산서 발행은 담당자 이메일주소, 전화번호, 계산서 발행처를 기입해주시면 바로 발행이 가능합니다. 그 외 서류수정, 재발행요청 등의 문의사항이 있으시면 경영지원팀으로 연락주세요. (직통전화 02-465-6236)
-
국민은행 : 016-25-0013-786
기업은행 : 023-064957-04-011
농 협 : 029-01-174763
우리은행 : 514-394506-13-001
예금주명 (주) 코스모진텍 -
저희 쪽에서 비밀번호 확인은 불가능하며 변경만 가능합니다. 대표전화 (02-922-7307)로 연락을 주시면 임시 비밀번호를 부여해 드리겠습니다. 부여받은 비밀번호는 차후에 변경 해주세요.
-
본 사이트는 Chrome, Safari, Firefox 및 IE 10 이상의 최신 브라우저에 최적화되어 있습니다.
HTML5, CSS3 기반으로 제작되어 IE 9 버전 이하에서는 일부 기능이 정상적으로 작동하지 않습니다.
Cosmogenetech 서비스를 100% 이용하기 위해선 IE 최신 버전을 다운로드 받으시거나
Chrome 등 다른 브라우저를 이용하시길 권장합니다.
* Chrome 다운받기
* IE 최신버전 다운받기

-
- 대표이사 : 이영준
- 사업자등록번호 : 209-81-25456
- 본사(04784)서울특별시 성동구 성수이로 10길 14 (성수동2가, 에이스 하이엔드 성수타워) 304~311호
- 대전(34166)대전광역시 유성구 유성대로 794(지번주소:장대동 342-1)6층 뉴토피아빌딩 601호
- Copyright ⓒ Cosmogenetech co, Ltd. All rights reserved
-
- 학술마케팅 (서울) 02-465-6217 02-499-3084 support@cosmogenetech.com
- 학술마케팅 (대전) 070-4771-7694 042-867-7301
- Sequencing (서울) 02-465-6265 02-921-3084 sequencing@cosmogenetech.com
- Sequencing (대전) 042-826-6265 042-867-7301 sequencing@cosmogenetech.com
- 미생물유전체 042-364-7301 042-867-7301 mgb@cosmogenetech.com
- Cloning 02-465-6216 02-921-3084 cloning@cosmogenetech.com
- Gene Synthesis 02-6457-6230 02-921-3084 genesynthesis@cosmogenetech.com
- LaboPass 02-465-6215 02-921-3084 labopass@cosmogenetech.com
- 연구개발 02-465-6277 02-6004-6278 rnd@cosmogenetech.com
- 대전 042-867-7308 042-867-7301
-
ARS
1번 DNA 시퀀싱 2번 올리고 주문배송 3번 클로닝/실험서비스 4번 제품 관련 5번 계산서발행/결제 6번 학술상담 7번 총무/회계/기타 0번 다시듣기
-